| (Nie pokazano 12 pośrednich wersji utworzonych przez tego samego użytkownika) | |||
| Linia 1: | Linia 1: | ||
==Rodopsyna== | ==Rodopsyna== | ||
| − | |||
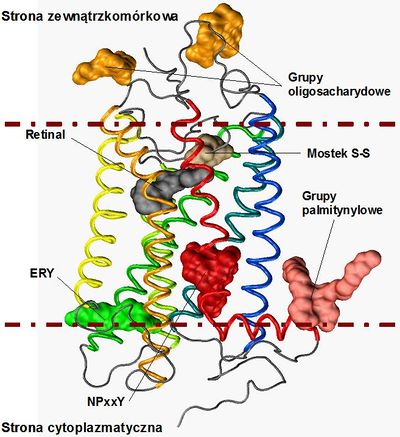
| − | + | [[Image:rodopsyna.jpg|thumb|upright|400px| Rys. 1. Mikrodomeny obecne w krystalicznej strukturze rodopsyny (PDB ID: 1U19) [10]. Helisy transmembranowe od TNH:I do TMH:VII (oraz H:VIII) zostały oznaczone kolorami od niebieskiego do czerwonego. Przerywane linie obrazują położenie granic błony komórkowej.]] | |
| − | Rodopsyna jest białkiem o masie 40 kDa (kilodaltonów), składającym się z 348 aminokwasów (AA). Rodopsyna, podobnie jak inne receptory GPCR, posiada charakterystyczny motyw siedmiu | + | Rodopsyna jest białkiem fotoreceptorowym zlokalizowanym w komórkach pręcikowych siatkówki oka. Receptor ten bierze udział w kaskadach sygnalizacyjnych umożliwiających przekształcenie sygnału świetlnego w impuls nerwowy [1]. Absorpcja pojedynczego fotonu jest wystarczająca do przejścia receptora w stan aktywny, co umożliwia związanie i aktywację białka G (transducyny) po wewnętrznej stronie błony komórkowej [2]. Aktywna podjednostka G<sub>α</sub> aktywuje fosfodiesterazę [3, 4], enzym hydrolizujący cykliczny GMP (cGMP) do GMP, co zmniejsza stężenie cGMP. Zmiana stężenia cGMP skutkuje czasowym zamknięciem kanałów potasowych powodując depolaryzację błony i przepływ prądu do synapsy i dalej do mózgu. Przekazany sygnał ulega istotnemu wzmocnieniu. Dzieję się tak gdyż pojedyncza molekuła rodopsyny może aktywować ok. 500 cząsteczek białka G. |
| + | |||
| + | Rodopsyna zbudowana jest z części białkowej opsyny oraz grupy prostetycznej 11-cis-retinalu (pochodnej witaminy A) (Rys. 1.). Kowalencyjnie związany ligand znajduje się w kieszeni hydrofobowej, otoczonej przez helisy (TMH:II do TMH:VII), którą przykrywa druga pętla cytoplazmatyczna (EL:II). Połączenie pomiędzy grupą aldehydową 11-cis-retinalu oraz łańcuchem bocznym Lys:7.43 tworzy uprotonowana zasada Schiffa [5]. Absorpcja kwantu światła przez 11-cis-retinal skutkuje jego izomeracją i przejściem w całkowicie-trans-retinal [1]. Zmiana w strukturze przestrzennej tego liganda prowadzi do zmian konformacyjnych całego receptora oraz jego aktywacji [6]. | ||
| + | |||
| + | Rodopsyna jest białkiem o masie 40 kDa (kilodaltonów), składającym się z 348 aminokwasów (AA). Rodopsyna, podobnie jak inne receptory [[GPCR]], posiada charakterystyczny motyw siedmiu α-helis przechodzących przez dwuwarstwę lipidową. Ich długość wynosi odpowiednio od 19 AA do 34 AA. Najkrótsza helisa VIII położona jest równolegle do dwuwarstwy lipidowej, po stronie cytoplazmatycznej [7]. | ||
| − | N-koniec rodopsyny zbudowany jest z 33 AA i jest dosyć sztywny bowiem jego konformację stabilizuje | + | [[Image:rodopsyna2.jpg|thumb|upright|400px| Rys. 2. Mikrodomeny obecne w krystalicznej strukturze rodopsyny (PDB ID: 1U19) [124]. Helisy transmembranowe od TNH:I do TMH:VII (oraz H:VIII) zostały oznaczone kolorami od niebieskiego do czerwonego. Przerywane linie obrazują położenie granic błony komórkowej.]] |
| + | |||
| + | N-koniec rodopsyny zbudowany jest z 33 AA i jest dosyć sztywny bowiem jego konformację stabilizuje β-kartka utworzona przez reszty Thr:4-Glu:5 oraz Tyr:10-Val:11 [8]. Dwie reszty asparaginowe zlokalizowane na pozycji 2 oraz 15 (Asn:2 oraz Asn:15) są miejscami glikozylacji receptora. Helisy transbłonowe rodopsyny posiadają nieregularną budowę oraz są odchylone od pionowej osi białka [7]. Zgięcia helis powstają głównie na skutek występujących w sekwencji reszt: prolin oraz glicyn. Największe zgięcia w TMH:VI oraz TMH:VII spowodowane są przez obecność dobrze zachowanych w sekwencji reszt Pro:6.50 oraz Pro:7.50. W helisach TMH:II oraz TMH:V występują odstępstwa od α-helikalnej struktury drugorzędowej na skutek obecności dodatkowych aminokwasów, odpowiednio: Gly:2.56 oraz Phe:5:47, tworzących w miejscu występowania fragmenty π-helisy. Również w rejonie zgięcia TMH:VII, fragment łańcucha białkowego przyjmuje nietypową konformację helisy 310. Pętle łączące helisy są relatywnie krótkie z wyjątkiem EL:II oraz IL:II. Najdłuższa pętla rodopsyny (EL:II), zlokalizowana pomiędzy helisami TMH:IV oraz TMH:V, szczelnie przykrywa miejsce wiązania retinalu zapobiegając prawdopodobnie samoistnej aktywacji rodopsyny, co jest niezwykle istotne dla procesu widzenia [9]. Fragmenty pętli łączącej TMH:IV oraz TMH:V: Ile:179 do Glu:181 oraz Ser:186 do Ile:189, tworzą strukturę β-kartki [8]. Za stabilizację pętli EL:II odpowiada dodatkowo wiązanie disulfidowe tworzone pomiędzy Cys:3.25 z helisy TMH:III oraz Cys:187 z pętli EL:II. Najdłuższą pętlą znajdującą się po stronie wewnątrzkomórkowej jest IL:II, łącząca TMH:IV oraz TMH:V. Porównanie wszystkich dostępnych struktur krystalicznych rodopsyny, zdeponowanych w bazie PDB, pozwala przypuszczać, iż fragment ten nie przyjmuje jednoznacznie określonej, stabilnej konformacji. Duże wartości czynnika temperaturowego dla tego odcinka łańcucha peptydowego również sugerują jego wysoką mobilność. Domena tworząca C-koniec receptora, od Asn:310 do Ala:348, nie jest dobrze upakowana. Między obecnymi tam aminokwasami nie występują żadne oddziaływania usztywniające ten fragment łańcucha białkowego. Krótka helisa H:VIII, znajdująca się na powierzchni błony, jest stabilizowana dzięki palmitylacji Cys:323 i Cys324 [10]. Długie palmitylowe łańcuchy, obecne na końcu tej helisy, są trwale zakotwiczone w błonie komórkowej. | ||
| + | |||
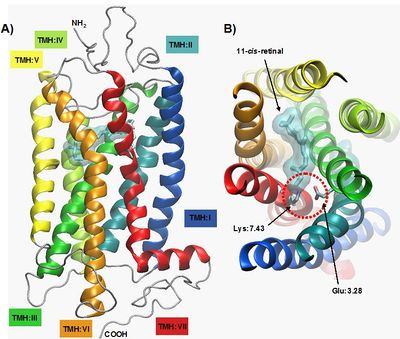
| + | W sekwencji rodopsyny występują charakterystyczne motywy dobrze zachowane w procesie ewolucji także w innych typach receptorów GPCR z rodziny A (Rys. 2.) [1, 11, 12]. Po stronie cytoplazmatycznej helisy TMH:III, znajduje się mikrodomena D(E)RY (a szczególnie środkowa, najbardziej zachowana w ewolucji reszta Arg:3.50), która tworzy połączenie pomiędzy helisami TMH:III i TMH:VI [11] poprzez utworzenie mostka solnego pomiędzy resztami Arg:3.50 oraz Glu:6.30. Podczas aktywacji receptora mostek ten ulega zerwaniu a część cytoplazmatyczna helisy TMH:VI ulega przemieszczeniu, tworząc w ten sposób miejsce na związanie białka G. Innym motywem o istotnym znaczeniu jest mikrodomena NPxxY obecna na helisie VII, która łączy helisy TMH:VII i H:VIII. Ten fragment łańcucha białkowego, również bierze udział w procesie aktywacji receptora prowadzącej do tworzenia kompleksu z białkiem G [2, 12]. Wzajemne położenie siedmiu helis transbłonowych rodopsyny stabilizowane jest dodatkowo przez oddziaływania hydrofobowe, jonowe, a także liczne wiązania wodorowe [1]. Badania krystalograficzne ujawniły także obecność cząsteczek wody w strukturze rodopsyny, które także uczestniczą w sieci wiązań wodorowych we wnętrzu receptora [13] i pośredniczą w procesie aktywacji. | ||
| + | |||
| + | == Zobacz też == | ||
| + | * [[GPCR]] | ||
| + | * [[GPCR – struktury krystaliczne]] | ||
| + | * [[Białko G ]] | ||
| + | * [[Receptory ]] | ||
| + | * [[Receptory błonowe ]] | ||
| + | * [[Receptory serotoninowe ]] | ||
| − | + | ==Literatura== | |
| + | # Filipek, S., et al., G protein-coupled receptor rhodopsin: a prospectus. Annu Rev Physiol, 2003. 65: p. 851-79. | ||
| + | # Filipek, S., et al., A concept for G protein activation by G protein-coupled receptor dimers: the transducin/rhodopsin interface. Photochem Photobiol Sci, 2004. 3(6): p. 628-38. | ||
| + | # Okada, T. and K. Palczewski, Crystal structure of rhodopsin: implications for vision and beyond. Curr Opin Struct Biol, 2001. 11(4): p. 420-6. | ||
| + | # Jang, G.F., et al., Mechanism of rhodopsin activation as examined with ring-constrained retinal analogs and the crystal structure of the ground state protein. J Biol Chem, 2001. 276(28): p. 26148-53. | ||
| + | # Janz, J.M. and D.L. Farrens, Role of the retinal hydrogen bond network in rhodopsin Schiff base stability and hydrolysis. J Biol Chem, 2004. 279(53): p. 55886-94. | ||
| + | # Grobner, G., et al., Observations of light-induced structural changes of retinal within rhodopsin. Nature, 2000. 405(6788): p. 810-3. | ||
| + | # Palczewski, K., et al., Crystal structure of rhodopsin: A G protein-coupled receptor. Science, 2000. 289(5480): p. 739-45. | ||
| + | # Filipek, S., et al., The crystallographic model of rhodopsin and its use in studies of other G protein-coupled receptors. Annu Rev Biophys Biomol Struct, 2003. 32: p. 375-97. | ||
| + | # Bourne, H.R. and E.C. Meng, Structure. Rhodopsin sees the light. Science, 2000. 289(5480): p. 733-4. | ||
| + | # Okada, T., et al., The retinal conformation and its environment in rhodopsin in light of a new 2.2 A crystal structure. J Mol Biol, 2004. 342(2): p. 571-83. | ||
| + | # Rovati, G.E., V. Capra, and R.R. Neubig, The highly conserved DRY motif of class A G protein-coupled receptors: beyond the ground state. Mol Pharmacol, 2007. 71(4): p. 959-64. | ||
| + | # Fritze, O., et al., Role of the conserved NPxxY(x)5,6F motif in the rhodopsin ground state and during activation. Proc Natl Acad Sci U S A, 2003. 100(5): p. 2290-5. | ||
| + | # Okada, T., et al., Functional role of internal water molecules in rhodopsin revealed by X-ray crystallography. Proc Natl Acad Sci U S A, 2002. 99(9): p. 5982-7. | ||
Aktualna wersja na dzień 02:29, 27 sty 2012
Rodopsyna
Rodopsyna jest białkiem fotoreceptorowym zlokalizowanym w komórkach pręcikowych siatkówki oka. Receptor ten bierze udział w kaskadach sygnalizacyjnych umożliwiających przekształcenie sygnału świetlnego w impuls nerwowy [1]. Absorpcja pojedynczego fotonu jest wystarczająca do przejścia receptora w stan aktywny, co umożliwia związanie i aktywację białka G (transducyny) po wewnętrznej stronie błony komórkowej [2]. Aktywna podjednostka Gα aktywuje fosfodiesterazę [3, 4], enzym hydrolizujący cykliczny GMP (cGMP) do GMP, co zmniejsza stężenie cGMP. Zmiana stężenia cGMP skutkuje czasowym zamknięciem kanałów potasowych powodując depolaryzację błony i przepływ prądu do synapsy i dalej do mózgu. Przekazany sygnał ulega istotnemu wzmocnieniu. Dzieję się tak gdyż pojedyncza molekuła rodopsyny może aktywować ok. 500 cząsteczek białka G.
Rodopsyna zbudowana jest z części białkowej opsyny oraz grupy prostetycznej 11-cis-retinalu (pochodnej witaminy A) (Rys. 1.). Kowalencyjnie związany ligand znajduje się w kieszeni hydrofobowej, otoczonej przez helisy (TMH:II do TMH:VII), którą przykrywa druga pętla cytoplazmatyczna (EL:II). Połączenie pomiędzy grupą aldehydową 11-cis-retinalu oraz łańcuchem bocznym Lys:7.43 tworzy uprotonowana zasada Schiffa [5]. Absorpcja kwantu światła przez 11-cis-retinal skutkuje jego izomeracją i przejściem w całkowicie-trans-retinal [1]. Zmiana w strukturze przestrzennej tego liganda prowadzi do zmian konformacyjnych całego receptora oraz jego aktywacji [6].
Rodopsyna jest białkiem o masie 40 kDa (kilodaltonów), składającym się z 348 aminokwasów (AA). Rodopsyna, podobnie jak inne receptory GPCR, posiada charakterystyczny motyw siedmiu α-helis przechodzących przez dwuwarstwę lipidową. Ich długość wynosi odpowiednio od 19 AA do 34 AA. Najkrótsza helisa VIII położona jest równolegle do dwuwarstwy lipidowej, po stronie cytoplazmatycznej [7].
N-koniec rodopsyny zbudowany jest z 33 AA i jest dosyć sztywny bowiem jego konformację stabilizuje β-kartka utworzona przez reszty Thr:4-Glu:5 oraz Tyr:10-Val:11 [8]. Dwie reszty asparaginowe zlokalizowane na pozycji 2 oraz 15 (Asn:2 oraz Asn:15) są miejscami glikozylacji receptora. Helisy transbłonowe rodopsyny posiadają nieregularną budowę oraz są odchylone od pionowej osi białka [7]. Zgięcia helis powstają głównie na skutek występujących w sekwencji reszt: prolin oraz glicyn. Największe zgięcia w TMH:VI oraz TMH:VII spowodowane są przez obecność dobrze zachowanych w sekwencji reszt Pro:6.50 oraz Pro:7.50. W helisach TMH:II oraz TMH:V występują odstępstwa od α-helikalnej struktury drugorzędowej na skutek obecności dodatkowych aminokwasów, odpowiednio: Gly:2.56 oraz Phe:5:47, tworzących w miejscu występowania fragmenty π-helisy. Również w rejonie zgięcia TMH:VII, fragment łańcucha białkowego przyjmuje nietypową konformację helisy 310. Pętle łączące helisy są relatywnie krótkie z wyjątkiem EL:II oraz IL:II. Najdłuższa pętla rodopsyny (EL:II), zlokalizowana pomiędzy helisami TMH:IV oraz TMH:V, szczelnie przykrywa miejsce wiązania retinalu zapobiegając prawdopodobnie samoistnej aktywacji rodopsyny, co jest niezwykle istotne dla procesu widzenia [9]. Fragmenty pętli łączącej TMH:IV oraz TMH:V: Ile:179 do Glu:181 oraz Ser:186 do Ile:189, tworzą strukturę β-kartki [8]. Za stabilizację pętli EL:II odpowiada dodatkowo wiązanie disulfidowe tworzone pomiędzy Cys:3.25 z helisy TMH:III oraz Cys:187 z pętli EL:II. Najdłuższą pętlą znajdującą się po stronie wewnątrzkomórkowej jest IL:II, łącząca TMH:IV oraz TMH:V. Porównanie wszystkich dostępnych struktur krystalicznych rodopsyny, zdeponowanych w bazie PDB, pozwala przypuszczać, iż fragment ten nie przyjmuje jednoznacznie określonej, stabilnej konformacji. Duże wartości czynnika temperaturowego dla tego odcinka łańcucha peptydowego również sugerują jego wysoką mobilność. Domena tworząca C-koniec receptora, od Asn:310 do Ala:348, nie jest dobrze upakowana. Między obecnymi tam aminokwasami nie występują żadne oddziaływania usztywniające ten fragment łańcucha białkowego. Krótka helisa H:VIII, znajdująca się na powierzchni błony, jest stabilizowana dzięki palmitylacji Cys:323 i Cys324 [10]. Długie palmitylowe łańcuchy, obecne na końcu tej helisy, są trwale zakotwiczone w błonie komórkowej.
W sekwencji rodopsyny występują charakterystyczne motywy dobrze zachowane w procesie ewolucji także w innych typach receptorów GPCR z rodziny A (Rys. 2.) [1, 11, 12]. Po stronie cytoplazmatycznej helisy TMH:III, znajduje się mikrodomena D(E)RY (a szczególnie środkowa, najbardziej zachowana w ewolucji reszta Arg:3.50), która tworzy połączenie pomiędzy helisami TMH:III i TMH:VI [11] poprzez utworzenie mostka solnego pomiędzy resztami Arg:3.50 oraz Glu:6.30. Podczas aktywacji receptora mostek ten ulega zerwaniu a część cytoplazmatyczna helisy TMH:VI ulega przemieszczeniu, tworząc w ten sposób miejsce na związanie białka G. Innym motywem o istotnym znaczeniu jest mikrodomena NPxxY obecna na helisie VII, która łączy helisy TMH:VII i H:VIII. Ten fragment łańcucha białkowego, również bierze udział w procesie aktywacji receptora prowadzącej do tworzenia kompleksu z białkiem G [2, 12]. Wzajemne położenie siedmiu helis transbłonowych rodopsyny stabilizowane jest dodatkowo przez oddziaływania hydrofobowe, jonowe, a także liczne wiązania wodorowe [1]. Badania krystalograficzne ujawniły także obecność cząsteczek wody w strukturze rodopsyny, które także uczestniczą w sieci wiązań wodorowych we wnętrzu receptora [13] i pośredniczą w procesie aktywacji.
Zobacz też
Literatura
- Filipek, S., et al., G protein-coupled receptor rhodopsin: a prospectus. Annu Rev Physiol, 2003. 65: p. 851-79.
- Filipek, S., et al., A concept for G protein activation by G protein-coupled receptor dimers: the transducin/rhodopsin interface. Photochem Photobiol Sci, 2004. 3(6): p. 628-38.
- Okada, T. and K. Palczewski, Crystal structure of rhodopsin: implications for vision and beyond. Curr Opin Struct Biol, 2001. 11(4): p. 420-6.
- Jang, G.F., et al., Mechanism of rhodopsin activation as examined with ring-constrained retinal analogs and the crystal structure of the ground state protein. J Biol Chem, 2001. 276(28): p. 26148-53.
- Janz, J.M. and D.L. Farrens, Role of the retinal hydrogen bond network in rhodopsin Schiff base stability and hydrolysis. J Biol Chem, 2004. 279(53): p. 55886-94.
- Grobner, G., et al., Observations of light-induced structural changes of retinal within rhodopsin. Nature, 2000. 405(6788): p. 810-3.
- Palczewski, K., et al., Crystal structure of rhodopsin: A G protein-coupled receptor. Science, 2000. 289(5480): p. 739-45.
- Filipek, S., et al., The crystallographic model of rhodopsin and its use in studies of other G protein-coupled receptors. Annu Rev Biophys Biomol Struct, 2003. 32: p. 375-97.
- Bourne, H.R. and E.C. Meng, Structure. Rhodopsin sees the light. Science, 2000. 289(5480): p. 733-4.
- Okada, T., et al., The retinal conformation and its environment in rhodopsin in light of a new 2.2 A crystal structure. J Mol Biol, 2004. 342(2): p. 571-83.
- Rovati, G.E., V. Capra, and R.R. Neubig, The highly conserved DRY motif of class A G protein-coupled receptors: beyond the ground state. Mol Pharmacol, 2007. 71(4): p. 959-64.
- Fritze, O., et al., Role of the conserved NPxxY(x)5,6F motif in the rhodopsin ground state and during activation. Proc Natl Acad Sci U S A, 2003. 100(5): p. 2290-5.
- Okada, T., et al., Functional role of internal water molecules in rhodopsin revealed by X-ray crystallography. Proc Natl Acad Sci U S A, 2002. 99(9): p. 5982-7.